Arbeitsgruppe Molekulare Mikrobiologie und Bioinformatik
Die Gesamtheit der Mikroorganismen, die einen Wirt besiedeln, wird als das Mikrobiom des Wirtes bezeichnet. Dieses Mikrobiom ist unterteilt in verschiedene Subpopulationen, die mit bestimmten Körperregionen und Organen in Verbindung stehen und als Mikrobiome dieser Regionen und Organe bezeichnet werden.
Hierbei liegen für unterschiedliche Wirte und Organe Unterschiede in der Zusammensetzung und in den Aufgaben der verschiedenen Mikrobiome vor. Zum Beispiel setzt sich das funktionelle Mikrobiom des Pansens bei Rindern aus einem Konsortium aus cellulytischen Pilzen und Protozoen, verschiedenen Bakteriengruppen und methanogenen Archaeen zusammen, während ein typisches Darmmikrobiom eines Monogastriers durch Bakterien charakterisiert ist.
Im Zusammenhang mit der Zusammensetzung eines Mikrobioms stehen auch seine Fähigkeiten. So besitzt jeder Mikroorganismus verschiedene spezifische Eigenschaften, die sich in einem Mikrobiom mit den Eigenschaften der anderen Mikroorganismen ergänzen und überlagern. Aus dieser Gesamtheit an Fähigkeiten ergibt sich zum Beispiel welche Leistungen ein Mikrobiom in der Umsetzung von Nahrung einbringen können und welche Interaktionen zwischen Mikrobiom und Wirt vorliegen.
Bei solchen Interaktionen kann es sich um gewünschte und unerwünschte Effekte handeln. Zum Beispiel wird die Ausbildung des Immunsystems auf der einen Seite durch die Anwesenheit von Mikroorganismen unterstützt und es wird eine Akzeptanz für förderlicher Mikroorganismen etabliert. Auf der anderen Seite können Störungen in der Zusammensetzung des Mikrobioms oder Ansiedlungen von pathogenen Mikroorganismen zu veterinärmedizinisch relevanten Befunden führen.
Um aus der Tierernährung resultierende Mikrobiom-assoziierte Fragestellungen zu adressieren, werden molekularbiologische, bioinformatische und statistische Methoden kombiniert:
- Sequenzierung von Markergenen aus Mikroorganismen in Kooperation mit anderen Instituten des FLI
- Identifizierung von Mikroorganismen basierend auf DNA-Sequenzen
- Analysen von Mikrobiomzusammensetzungen basierend auf klassischen ökologischen Maßeinheiten
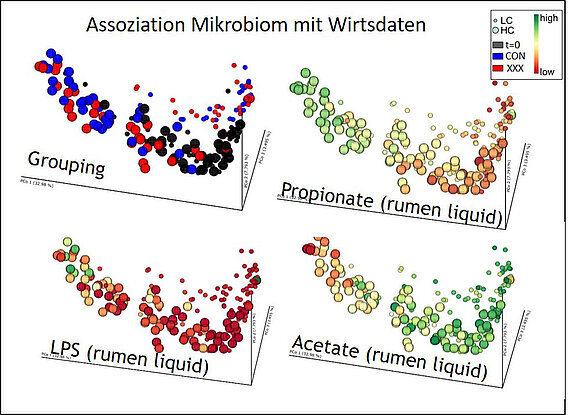
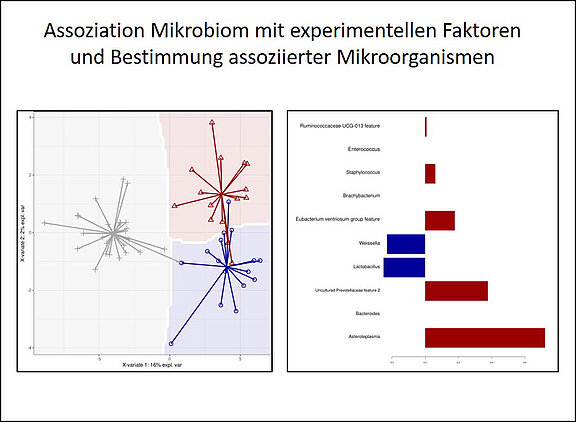
- Assoziation von Mikroorganismengruppen mit Beobachtungen aus dem Wirt
Neben der Anwendung von molekularbiologischen und bioinformatischen Methoden im Rahmen von Fragestellungen, die mit Mikrobiomen in Verbindung stehen, werden bioinformatische und moderne informatische Methoden aus dem Bereich des maschinellen Lernens in zunehmenden Maße in der Beobachtung und Charakterisierung von Nutztieren eingesetzt.
So werden beispielsweise Ergebnisse aus Methoden zur Analyse von Genexpressionen mit Daten aus verschiedenen tierspezifischen Analysen in übergreifenden statistischen Verfahren kombiniert. Solche übergreifenden Analysen von Daten bieten eine Möglichkeit, ein relativ breites Bild des Gesundheitszustandes eines Tiers zu erhalten. Zusätzlich dienen Befunde aus übergreifenden Analysen als Grundlage für die Überprüfung von Ergebnissen maschineller Lernmodelle, welche in zunehmendem Maße zur automatisierten Beschreibung von Tierzuständen herangezogen werden können. Hierbei ist es ein besonderes Ziel, Daten aus verschiedenen Quellen, wie bildgebenden Verfahren, automatisiert aufzubereiten, um sie für automatisierte maschinelle Lernmethoden nutzbar zu machen.