Hintergrund:
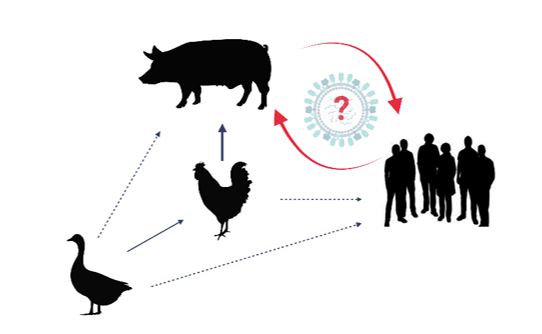
Abgesehen von der aktuellen Pandemie verusacht durch das Virus SARS-CoV-2, wurden alle Pandemien der letzten hundert Jahre ausnahmslos von Influenzaviren verursacht. Um Pandemien besser zu verstehen und im Sinne der „One Health“ Forschung entstand dieses Projekt zur Untersuchung des Übergangs von Influenzaviren zwischen Menschen und Schweinen.
„Schweinegrippe“ wurde seit dem globalen Ausbruch 2009 weltweit bekannt. Der Herd dieser Pandemie lag vermutlich in Südamerika, wobei sich Segmente von Influenzaviren aviären, humanen und porcinen Ursprungs im Schwein vermischten und eine weltweite Verbreitung des Virus in der menschlichen Bevölkerung verursachte.
Durch den engen Kontakt zu Menschen und der hohen Populationsdichte in heutigen Haltungsformen gelten Schweine als wichtiges Reservoir und „Mischgefäß“ diverser Influenzaviren mit möglicherweise präpandemischen Potential. Weltweit werden regelmäßig zoonotische Infektionen, verursacht durch Schweinegrippenviren, gemeldet. Auch in den Jahren 2021/22 wurden in Deutschland zwei Fälle porciner Influenza bei Menschen bestätigt.